近日,河南大学农学院林敏团队与中国农科院生物所相关团队合作,系统开展RNA结合蛋白Hfq介导的转录后调控机制研究,通过Hfq-RNA互作组和转录组整合分析,解析根际细菌施氏假单胞菌中参与固氮调控的复杂网络。取得的相关成果发表在微生物学期刊mSphere上。

在根际微生物中,Hfq作为一个全局性的转录后调控因子,直接或间接调控生长繁殖、趋化运动、环境适应和生物固氮等复杂生理生化过程。固氮施氏假单胞菌A1501是一株分离自水稻根际的模式联合固氮菌,在生产应用中其固氮活性和节肥增长能力受根际非生物胁迫因子影响较大。本团队在前期研究中发现,施氏假单胞菌hfq基因缺失突变株的固氮酶活、碳代谢利用、胞外多糖合成以及根际定殖等能力均明显下降,同时hfq缺失显著影响相关功能基因表达(PNAS, 2016; npj Biofilms and Microbiomes, 2021; iScience 2022),预测Hfq在固氮菌基因网络调控中发挥了核心作用,然而,由于传统方法学上的局限,目前关于Hfq直接结合的靶标RNA知之甚少,转录后相关网络调控机制尚不清楚。
本研究采用增强的紫外交联免疫沉淀(eCLIP)方法,并结合高通量测序技术,在全基因组水平上测定了Hfq直接结合的靶标RNA序列,比较了固氮和非固氮条件下的靶标RNA丰度差异。通过全基因组注释分析,鉴定了数百个此前从未发现的Hfq直接调控的靶标基因和一百多个功能未知的Hfq-靶标RNA结合序列,其中包括参与固氮、碳代谢、生物膜形成等重要生物过程的固氮基因nifM、铵载体基因amtB、外模蛋白oprB和趋化蛋白cheZ等,为今后开展根际固氮微生物环境适应及其与作物互作机制研究提供了新靶标与新方向。RNA组和转录组数据整合分析结果还表明,Hfq能够与其它代谢途径的调控蛋白,如次生代谢抑制蛋白RsmA、σ因子AlgU和固氮正调节蛋白NifA、非编码RsmY以及这些调控蛋白的某些靶标基因存在直接的相互作用,从而形成了一个转录后控制生物固氮的复杂调控网络,赋予了根际固氮菌A1501在竞争激烈的根际环境中具有的竞争优势。
本研究在全基因组水平上系统解析了Hfq介导的复杂调控网络,拓宽了根际固氮菌Hfq-RNA互作组内涵,为下一步深入研究根际环境中固氮菌-宿主作物互作网络调控机制研提供了一个新框架。同时,Hfq新靶标及其在调控网络中形成的新节点发现,将为固氮模块的智能设计,固氮底盘的系统优化和新一代根际联合固氮体系创建提供了重要的理论指导。
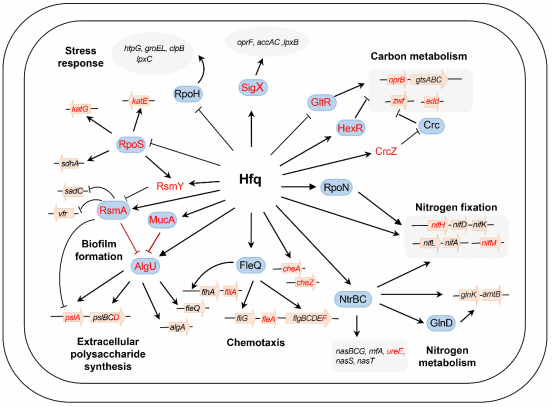
根际固氮微生物中Hfq介导的固氮与相关代谢途径互作调控网络
该研究得到国家重点研发计划合成生物学专项、国家自然科学基金重点项目和指南引导类原创探索计划项目的资助。河南大学农学院林敏教授为论文共同通讯作者,河南大学农学院冯海超研究员和生命科学学院魏金凤教授参与了此项工作。(作者:冯海超 审核:林敏)
Copyright © 2021 河南大学农学院 版权所有
